生化学的手法を用いたクロマグロ大西洋と太平洋系統の簡便・迅速な判別手法の開発
[要約]
クロマグロの大西洋と太平洋系統間で見られたミトコンドリアDNAの塩基配列差異の情報から、試験的に1ないし3個の塩基置換がある2つの領域を選び、これらの差異を利用した系統の判別手法について検討した。各系統標本についてこの2領域を含むDNA断片を蛍光色素を取り込ませてPCR増幅した。これら2領域について各系統ごとに相補的なDNAプローブを作成し、増幅DNA断片とこれらプローブ間の相補性を蛍光強度によって測定した。その結果、各系統と相補的なプローブと相補的でないプローブ間で有意な蛍光強度の違いが見られた。本手法は制限酵素処理や電気泳動を必要としないため、大幅な分析時間の短縮が可能である。
遠洋水産研究所 浮魚資源部 熱帯性まぐろ研究室
[連絡先]0543-36-6013
[推進会議]遠洋漁業
[専門]資源管理
[対象]まぐろ
[分類]研究
[背景・ねらい]
まぐろ類の種判別は生態研究に重要であるだけでなく、近年では原産地証明を必要とするクロマグロの違法輸入を抑止する上で必要となっている。大西洋と太平洋系統間にはミトコンドリアDNAの塩基配列に大きな差異があることがわかっており、PCR増幅したDNA断片を制限酵素処理し電気泳動することによって判別する手法は確立している。しかし、この手法では2日程度の時間が必要であり、現場での応用には不向きである。そこで、判別システムをさらに簡便・迅速なものとするために、制限酵素処理や電気泳動を省略できる新規手法の開発を目的とした。
[成果の内容・特徴]
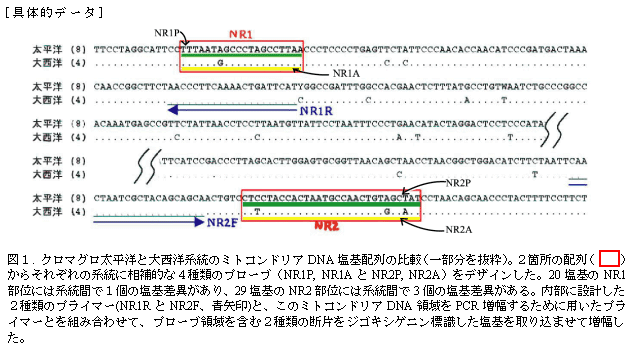
- クロマグロの大西洋と太平洋系統からそれぞれ4個体と8個体についてミトコンドリアDNAの部分領域の塩基配列を分析した(図1)。この配列情報から系統間で20塩基で1個の差異がみられる領域(NR1)と29塩基で3個の差異がみられる領域(NR2)を選び、大西洋と太平洋系統に相補的な1本鎖DNA(オリゴDNAプローブ)を4種類(NR1A、NR1P、NR2A、NR2P)デザインした。
- 系統間で差異がない領域において2種のPCRプライマー(NR1R、NR2F)を作製し(図1)、各系統標本においてNR1、NR2それぞれの領域を含むDNA断片を蛍光色素を取り込ませてPCR増幅した。
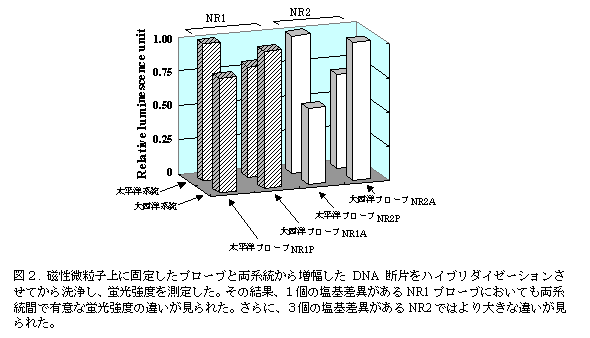
- 磁性微粒子上に4種のオリゴDNAプローブをそれぞれ固定し、各系統から増幅したDNA断片とハイブリダイゼーション(くっつける)させ洗浄した後、蛍光強度の違いを測定した(図2)。この原理は、オリゴDNAプローブと1つでも塩基の違いがある増幅DNA断片とではくっつく力が弱くなると予想されることである。すなわち、ある系統から増幅したDNA断片とDNAプローブの配列が一致していれば強くくっついており洗浄しても洗い流されないため蛍光色素がついている増幅DNA断片が量的に多く残るというわけである。
- 系統間で1個の塩基差異があるDNAプローブを用いた場合でも有意な蛍光強度の差異が見られ、3個の差異があるプローブを用いた場合には蛍光強度の差異がさらに大きくなった(図2)。
- 本手法は磁性微粒子を用いるため、ハイブリダイゼーションと洗浄が非常に簡便である。また、制限酵素処理と電気泳動が不要になり、分析時間が大幅に短縮できることがわかった。
[成果の活用面・留意点]
DNA抽出の自動化とPCR増幅の短縮及び、本手法を組み合わせれば、大西洋と太平洋のクロマグロ系統の判別は1~2時間程度でできるものと考えられる。本システムの小型化が図れれば、現場での簡便・迅速な判別に実用可能である。本研究ではクロマグロの2系統を標本として使用したが、他のまぐろ類のDNA情報を検討することによって、まぐろ類全種の判別も可能である。
[その他]
研究課題名:DNA分析によるまぐろ・かじき類の種判別とストック識別及び手法の簡便迅速化
研究期間:平成13~17年度
研究担当者:張 成年(企画連絡室)・松永 是、竹山春子(東京農工大)
発表論文等:Takeyama, H., Chow, S., Tsuzuki, H. and Matsunaga, T. (2001): Mitochondrial DNA sequence variation within and between Thunnus tuna species and its application to species identification. Journal of Fish Biology 58: 1646-1657.
Takeyama, H., Tsuzuki, H., Chow, S., Nakayama, H. and Matsunaga, T. (2000): Discrimination between Atlantic and Pacific subspecies of northern bluefin tuna (Thunnus thynnus) by magnetic-capture hybridization using bacterial
magnetic particles. Marine Biotechnology 2: 309-313.
Takeyama, H., Tsuzuki, H., Chow, S. and Matsunaga, T. (2000): Full-automated identification system of tuna species using species-specific oligonucleotide probe immobilized on bacterial magnetic particles. International Marine Biotechnology Conference 2000 p. 179. (Townsville, Australia).


